北理工課題組在蛋白質(zhì)與RNA相互作用領(lǐng)域發(fā)表綜述文章
發(fā)布日期:2024-07-26 供稿:生命學(xué)院 攝影:生命學(xué)院
編輯:肖雯 審核:常非 閱讀次數(shù):
近日,北京理工大學(xué)生命學(xué)院的金花教授團(tuán)隊(duì)在WIREs RNA(Wiley Interdisciplinary Reviews - RNA)發(fā)表了題為“Revealing the hidden RBP-RNA interactions with RNA modification enzyme-based strategies”的綜述。文章綜述了RNA修飾酶的特點(diǎn)和基于RNA修飾酶開發(fā)的蛋白質(zhì)與RNA互作的研究方法。該工作以金花教授和博士生李沖為共同第一作者,樸威蘭博士為通訊作者,北京理工大學(xué)為第一通訊單位。
RNA結(jié)合蛋白(RNA-binding proteins,RBPs)是生物體內(nèi)強(qiáng)有力的多功能調(diào)節(jié)因子,通過在多個(gè)水平上調(diào)節(jié)基因表達(dá),在生物體發(fā)育、代謝和各種疾病中發(fā)揮重要作用。對(duì)RBP功能深入研究的需求,推動(dòng)了RBP-RNA相互作用檢測(cè)方法的快速發(fā)展。近年來,將RNA修飾酶(RNA modification enzymes,RME)與感興趣的RBP融合的檢測(cè)方法開發(fā)成為一個(gè)熱門研究方向。該文章綜述了包括作用于RNA的腺嘌呤脫氨基酶ADAR、末端核苷酸轉(zhuǎn)移酶TENT和激活誘導(dǎo)的胞嘧啶脫氨酶/載脂蛋白B mRNA編輯酶催化多肽樣(AID/APOBEC)蛋白家族脫氨酶在內(nèi)的,RNA修飾酶的生物學(xué)功能、生物化學(xué)活性和來自于酶自身與其結(jié)構(gòu)域和伴侶蛋白的底物特異性。同時(shí),討論了RNA修飾酶活性篩選系統(tǒng),以及具有工程酶活性的RNA修飾酶突變。此外,文章提供了基于RNA修飾酶和基于交聯(lián)和免疫純化(CLIP)的RBP靶標(biāo)檢測(cè)策略的基本原理、優(yōu)缺點(diǎn)和應(yīng)用的系統(tǒng)概述,包括基于A-G的RNA編輯酶鑒定RNA結(jié)合蛋白靶標(biāo)的TRIBE(Targets of RNA-binding proteins Identified By Editing)、基于RNA加尾酶的方法RNA Tagging、C-U的RNA編輯酶APOBEC1介導(dǎo)的編輯靶標(biāo)分析技術(shù)STAMP(Surveying Targets by APOBEC-Mediated Profiling)、CLIP-seq及其衍生技術(shù) (圖1)。
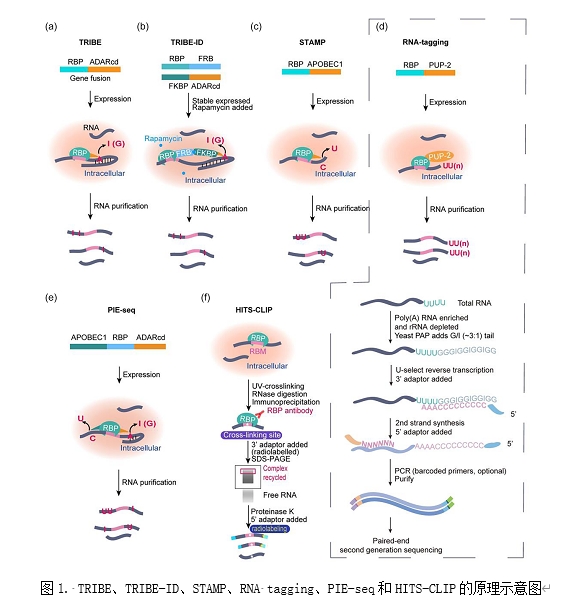
這篇綜述指出經(jīng)典的CLIP方法存在抗體依賴性、假陽(yáng)性高、重復(fù)性低的弊端,即使是改進(jìn)后的iCLIP技術(shù)仍存在實(shí)驗(yàn)操作繁瑣的缺點(diǎn),經(jīng)歷了十幾年的優(yōu)化過程后,CLIP衍生技術(shù)走向了成熟。CLIP的改進(jìn)版本構(gòu)建了上百種RBP和RNA互作數(shù)據(jù)庫(kù),并且完成了高通量檢測(cè)方法ABC的開發(fā)。同時(shí),文章也指出RBP-RME方法可以檢測(cè)細(xì)胞特異性的、不同種類和不同亞細(xì)胞結(jié)構(gòu)RNA靶標(biāo)的特點(diǎn),并且具有同源異構(gòu)體RNA(isoform)敏感性,這使得RBP-RME方法如TRIBE/STAMP具有潛在的強(qiáng)大功能,進(jìn)一步奠定了RBP-RME方法的基石地位。文章首次提出RBP-RME方法的應(yīng)用規(guī)則和測(cè)序方法選擇指南,并且在CLIP-seq為基礎(chǔ)的方法和RBP-RME方法兩者的選擇中給出具有參考價(jià)值的建議。根據(jù)這篇論文,人們可以洞察RNA修飾酶及RBP-RME方法的未來應(yīng)用和改進(jìn)方向。
遺傳與病理實(shí)驗(yàn)室長(zhǎng)期從事RNA編輯酶改造與檢測(cè)RBP-RNA互作方法的開發(fā)工作。2021年,在Science旗下的子刊Science Advances發(fā)表題為“TRIBE editing reveals specific mRNA targets of eIF4E-BP in Drosophila and in mammals”的研究論文,該論文在哺乳動(dòng)物中利用TRIBE的改進(jìn)版本HyperTRIBE證明,在果蠅和哺乳動(dòng)物體內(nèi)mTOR抑制條件下,eIF4E結(jié)合蛋白(4E-BP)通過eIF4E與一組特定的mRNA結(jié)合并抑制它們的翻譯。這些結(jié)果解釋了4E-BP如何優(yōu)先抑制具有TOP基序的mRNA子集的翻譯,為4E-BP和它的靶標(biāo)mRNA體內(nèi)互作提供了第一個(gè)體內(nèi)證據(jù)。2023年,課題組樸威蘭博士和博士生李沖為共同第一作者,在International Journal of Molecular Sciences發(fā)表題為“Identification of RNA-Binding Protein Targets with HyperTRIBE in Saccharomyces cerevisiae ”的研究論文。該文章通過將RBP融合到人類RNA編輯酶ADAR2的高活性催化結(jié)構(gòu)域,并在酵母細(xì)胞中表達(dá)融合蛋白,首次在酵母中建立了有效的HyperTRIBE技術(shù),鑒定了RNA結(jié)合蛋白KHD1p和BFR1p的RNA靶標(biāo)。
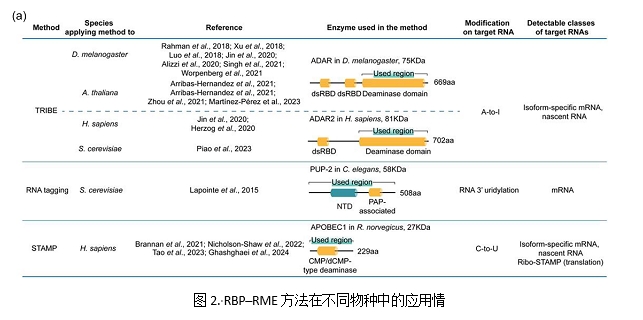
本實(shí)驗(yàn)室和其他研究團(tuán)隊(duì)的研究成果顯示,HyperTRIBE具有無(wú)需抗體、背景低、靈敏度高、重復(fù)性好、文庫(kù)制備過程簡(jiǎn)單等優(yōu)勢(shì),為釀酒酵母、哺乳動(dòng)物、果蠅和植物中RBP靶標(biāo)的鑒定提供了可靠的策略(圖2)。未來針對(duì)編輯酶的改造和其他RBP-RME方法的改進(jìn),將進(jìn)一步提高RBP的RNA靶標(biāo)鑒定效率并加深對(duì)生物學(xué)原理和人類健康與疾病的理解。
實(shí)驗(yàn)室相關(guān)研究由國(guó)家自然科學(xué)基金面上項(xiàng)目和中央高校基本科研業(yè)務(wù)費(fèi)專項(xiàng)資金資助,北京理工大學(xué)生物與醫(yī)學(xué)工程公共實(shí)驗(yàn)中心提供儀器、技術(shù)等支持。
全文鏈接:https://wires.onlinelibrary.wiley.com/doi/10.1002/wrna.1863
分享到:
